Confinés, nous suivons tous l’évolution de ce satané virus. Non ? Ce que je vous propose au travers de cet article c’est de récupérer par vous même les données officielles de Santé Publique ainsi que du ministère de la santé afin de faire vos propres analyses. Ces données sont déposées dans github : https://github.com/opencovid19-fr/data mais elles sont au format YAML et nécessitent quelques retouches pour être utilisable.
Format de données YAML
Ce format est un format assez simple d’utilisation, une sorte de json très simplifié qui ressemble à des données structurées via de l’indentation. Voici un exemple de fichier :
date: "2020-01-24"
source:
nom: Ministère des Solidarités et de la Santé
url: https://solidarites-sante.gouv.fr/IMG/pdf/200124-cp_coronavirus.pdf
archive: https://web.archive.org/web/20200322183046/https://solidarites-sante.gouv.fr/IMG/pdf/200124-cp_coronavirus.pdf
donneesNationales:
casConfirmes: 3
hospitalises: 3
deces: 0
donneesRegionales:
- nom: Île-de-France
code: REG-11
casConfirmes: 2
hospitalises: 2
- nom: Nouvelle-Aquitaine
code: REG-75
casConfirmes: 1
hospitalises: 1Heureusement, et comme souvent Python fournit une librairie pour gérer ce type de format automatiquement et le convertir dans un format utilisable simplement : JSON.
Pour ce faire, il faut installer le package :
pip install yamlEnsuite il suffira de lire le fichier avec la méthode :
json_data = yaml.load(, Loader=yaml.FullLoader)La valeur de json_data est retournée au format JSON.
Récupération des données brutes
Nous allons récupérer les données directement en interrogeant Github. Pour ce faire il faut interroger Github directement sur les données (raw) dans : https://raw.githubusercontent.com/opencovid19-fr/data/master/sante-publique-france/
Chaque fichier est au format YYYY-MM-DD.yaml il faudra donc balayer toutes les dates depuis le début jusqu’à aujourd’hui.
r = requests.get('https://raw.githubusercontent.com/opencovid19-fr/data/master/sante-publique-france/2020-03-24.yaml')
if (r.status_code==200):
print (yaml.load(r.text, Loader=yaml.FullLoader))Créer le nom de fichier à récupérer n’a rien de bien complexe, il suffit de récupérer toutes les dataes les unes après les autres et de recomposer le nom de fichier dans GitHub:
hier = date.today() + timedelta(-1)
shier = hier.strftime("%Y-%m-%d")
fichier = 'https://raw.githubusercontent.com/opencovid19-fr/data/master/sante-publique-france/' + shier + '.yaml'Si on veut récupérer le fichier d’hier, voici une petite fonction qui va le faire en quelques lignes :
def RecupCovid19Hier():
hier = date.today() + timedelta(-1)
shier = hier.strftime("%Y-%m-%d")
fichier = 'https://raw.githubusercontent.com/opencovid19-fr/data/master/sante-publique-france/' + shier + '.yaml'
out = ""
r = requests.get(fichier)
if (r.status_code==200):
out = yaml.load(r.text, Loader=yaml.FullLoader)
return outRécupérons les données san Santé Publique France
Les données ont été publiées à partir du 23 janvier 2020.
Voici la fonction qui va permettre de récupérer les données au travers des fichiers YAML (un par jour) via les principes expliqués ci-dessus.
import yaml
import requests
import time
from datetime import datetime, timedelta, date
import pandas as pd
# INITIALISATION DE VARIABLES GLOBALES
sDateDebut = "2020-01-23"
sAujourdhui = date.today().strftime("%Y-%m-%d")
dDemarrageDate = datetime.fromisoformat(sDateDebut)
cols = ['Date',
'FR Tot Cas Confirmés',
'FR Tot Décès',
'WW Tot Cas Confirmés',
'WW Tot Décès',
'Source',
'FR Tot Guéris',
'FR Tot Hospitalisés'
]
# FONCTIONS
def recupDataWithException2(_jsonout, _index1, _index2):
try:
data = _jsonout[_index1][_index2]
except:
data = 0
return data
def AjouteSantePubliqueFrLigne(jsonout, df):
df = df.append({
cols[0] : jsonout['date'] ,
cols[1] : recupDataWithException2(jsonout, 'donneesNationales', 'casConfirmes') ,
cols[2] : recupDataWithException2(jsonout, 'donneesNationales', 'deces'),
cols[3] : recupDataWithException2(jsonout,'donneesMondiales', 'casConfirmes'),
cols[4] : recupDataWithException2(jsonout,'donneesMondiales', 'deces'),
cols[5] : 'sante-publique-france',
cols[6] : 0,
cols[7] : 0
} ,
ignore_index=True)
return df
def ajouteLigneVide(df, madate):
df = df.append({
cols[0] : madate ,
cols[1] : 0,
cols[2] : 0,
cols[3] : 0,
cols[4] : 0,
cols[5] : 'no-data',
cols[6] : 0,
cols[7] : 0
} ,
ignore_index=True)
return df
# RECUPERATION DONNEES SANTE PUBLIQUE FRANCE
def RecupereDonneesSantePubliqueFrance():
print ('--> Démarrage du process')
sDateParcours = sDateDebut
i=1
requesReturnCode=200
yamls = pd.DataFrame()
while (sDateParcours != sAujourdhui):
myDate = dDemarrageDate + timedelta(days=i)
sDateParcours = myDate.strftime("%Y-%m-%d")
fichier = 'https://raw.githubusercontent.com/opencovid19-fr/data/master/sante-publique-france/' + sDateParcours + '.yaml'
#print ('--> sante-publique-france / ', sDateParcours)
req = requests.get(fichier)
requesReturnCode = req.status_code
if (requesReturnCode==200):
yamlout = yaml.load(req.text, Loader=yaml.FullLoader)
yamls = AjouteSantePubliqueFrLigne(yamlout, yamls)
else:
print ("(*) Pas de données pour ", myDate.strftime("%Y-%m-%d"))
# ajoute une ligne vide
yamls = ajouteLigneVide(yamls, myDate.strftime("%Y-%m-%d"))
i=i+1
print ('--> Fin du process')
return yamls
data = RecupereDonneesSantePubliqueFrance()
dataCette fonction retourne donc un dataframe Pandas qui contient les données de Santé Publique France.
Date FR Tot Cas Confirmés FR Tot Décès FR Tot Guéris FR Tot Hospitalisés Source WW Tot Cas Confirmés WW Tot Décès
0 2020-01-24 3.0 0.0 0.0 0.0 sante-publique-france 897.0 26.0
1 2020-01-25 3.0 0.0 0.0 0.0 sante-publique-france 1329.0 41.0
2 2020-01-26 3.0 0.0 0.0 0.0 sante-publique-france 2026.0 56.0
3 2020-01-27 3.0 0.0 0.0 0.0 sante-publique-france 2820.0 81.0
4 2020-01-28 4.0 0.0 0.0 0.0 sante-publique-france 0.0 0.0
... ... ... ... ... ... ... ... ...
64 2020-03-28 37575.0 2314.0 0.0 0.0 sante-publique-france 591971.0 27090.0
65 2020-03-29 0.0 0.0 0.0 0.0 no-data 0.0 0.0
66 2020-03-30 0.0 0.0 0.0 0.0 no-data 0.0 0.0
67 2020-03-31 0.0 0.0 0.0 0.0 no-data 0.0 0.0
68 2020-04-01 0.0 0.0 0.0 0.0 no-data 0.0 0.0
69 rows × 8 columnsRécupération des données du ministère de la santé
La récupération va suivre le même principe. Néanmoins ici on ne va pas créer un nouveau dataframe Pandas, nous allons plutôt enrichir les données déjà récupérées précédemment avec celles du ministère (on récupère notamment quelques informations supplémentaires comme le nombre de guéris, le nombre d’hospitalisation).
Voici la fonction qui récupère et enrichit le dataframe précédent :
# RECUPERATION DONNEES SANTE PUBLIQUE FRANCE
def RecupereDonneesMinistereSante(yamls):
i=1
sDateParcours = sDateDebut
print ('--> Démarrage du process')
while (sDateParcours != sAujourdhui):
myDate = dDemarrageDate + timedelta(days=i)
sDateParcours = myDate.strftime("%Y-%m-%d")
#print("Traintement date: ", dateParcours)
fichier = 'https://raw.githubusercontent.com/opencovid19-fr/data/master/ministere-sante/' + sDateParcours + '.yaml'
req = requests.get(fichier)
requesReturnCode = req.status_code
if (requesReturnCode==200):
yamlout = yaml.load(req.text, Loader=yaml.FullLoader)
if (yamls[cols[5]][i-1] == "no-data"):
print ("(*) Modifie les données manquantes pour la date du ", myDate.strftime("%Y-%m-%d"))
# Modifie toute la ligne
yamls.loc[i-1, cols[1]] = recupDataWithException2(yamlout, 'donneesNationales', 'casConfirmes')
yamls.loc[i-1, cols[2]] = recupDataWithException2(yamlout, 'donneesNationales', 'deces')
yamls.loc[i-1, cols[3]] = recupDataWithException2(yamlout, 'donneesMondiales', 'casConfirmes')
yamls.loc[i-1, cols[4]] = recupDataWithException2(yamlout, 'donneesMondiales', 'deces')
yamls.loc[i-1, cols[5]] = "ministere-sante"
# Ajoute les nouveaux champs
yamls.loc[i-1, cols[6]] = recupDataWithException2(yamlout, 'donneesNationales', 'hospitalises')
yamls.loc[i-1, cols[7]] = recupDataWithException2(yamlout, 'donneesNationales', 'gueris')
i=i+1
print ('--> Fin du process')
return yamlsQuelques finitions sur les données
Les données brutes récupérées sont des données de cumuls. Par ailleurs, on constate que certains jours n’ont pas été officiellements publiés (no-data). La conséquence est que les données présentent des « trous » ce qui est un peu genant pour des données de cumul. Nous allons combler ces trous :
def Bouchetrous(yamls):
# On reparcourre toutes les lignes déjà récupérées précédemment
i=1
for index, row in yamls.iterrows():
if (yamls[cols[5]][i-1] == "no-data"):
for j in [1, 2, 3, 4, 6, 7]:
yamls.loc[i-1, cols[j]] = yamls[cols[j]][i-2]
#yamls[cols[j]][i-1] = yamls[cols[j]][i-2]
i += 1
return yamlsPour ceux qui veulent aller plus loin , il pourrait être aussi intéressant de « desaggréger » les données, ou faire d’autres opérations.
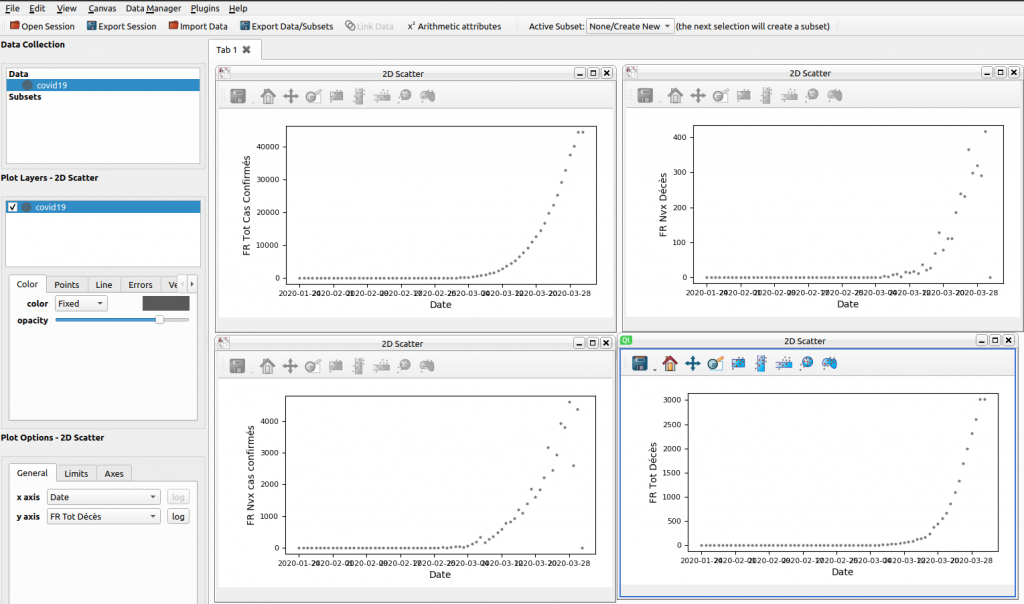
Le résultat ?
Voici un aperçu des données, qui malheureusement nous montrent que le pic de l’épidémie n’est pas encore atteint.
Vous pouvez récupérer le code complet sur GitHub.
Pour ceux que ça interresse je vais essayer de faire tourner la moulinette tous les jours et mettre les résutats dans un fichier csv sur GitHub.
Ingénieur en informatique avec plus de 20 ans d’expérience dans la gestion et l’utilisation de données, Benoit CAYLA a mis son expertise au profit de projets très variés tels que l’intégration, la gouvernance, l’analyse, l’IA, la mise en place de MDM ou de solution PIM pour le compte de diverses entreprises spécialisées dans la donnée (dont IBM, Informatica et Tableau). Ces riches expériences l’ont naturellement conduit à intervenir dans des projets de plus grande envergure autour de la gestion et de la valorisation des données, et ce principalement dans des secteurs d’activités tels que l’industrie, la grande distribution, l’assurance et la finance. Également, passionné d’IA (Machine Learning, NLP et Deep Learning), l’auteur a rejoint Blue Prism en 2019 et travaille aujourd’hui en tant qu’expert data/IA et processus. Son sens pédagogique ainsi que son expertise l’ont aussi amené à animer un blog en français (datacorner.fr) ayant pour but de montrer comment comprendre, analyser et utiliser ses données le plus simplement possible.


2 Replies to “Covid-19 : Récupérer les données brutes officielles”